研究人員
博士後研究學者
- 蔡郁偉 Yu-Wei Tsay
研究群介紹
本實驗室的研究是以資訊技術應用在生物和醫學研究為主,針對不同生物體學(omics)上的生物學和生物醫學進行解析,主要研究可分成基因體和轉譯體研究,蛋白體及代謝體研究兩大部份,分別詳述如下。(1) 基因體和轉譯體研究
處理巨量定序資料的方法論開發:
新世代定序技術已成為基因體和轉錄體研究的主要工具。然而定序的資料量相當龐大,定序的序列也常有錯誤,因此在資料處理時,常造成記憶體不足和計算時間冗長等困擾,亟待解決。因此,近年來我們致力於發展新的方法和開發新的工具來解決一連串計算的問題。在次世代 read mapping 上,我們發展了一個極為快速的演算法 Kart。Kart 將一個 read 分解成更小的碎片,並將其個別匹配到基因序列。實驗結果證實 Kart 比一般 mappers 快三到十倍之多,而且錯誤容忍度及準確度均極高。同樣的方法也可以應用在 RNA-seq 上,效果也甚佳。在基因體組裝方面,我們設計一套以延伸為基礎的組裝程式(稱為 JR-Assembler)利用整個短序做延伸,加快執行速度。另外利用跨過小於序列長度的重複片段等方式來大幅降低記憶體的需求和執行時間,同時也考慮組裝的品質。和其他程式比較,JR-Assembler 的記憶體使用率和執行時間更有效率,而且保持良好的組裝品質,特別對於序長度等於及大於 150 bp 的基因組資料。
處理巨量定序資料的工具和資訊整合平台開發:
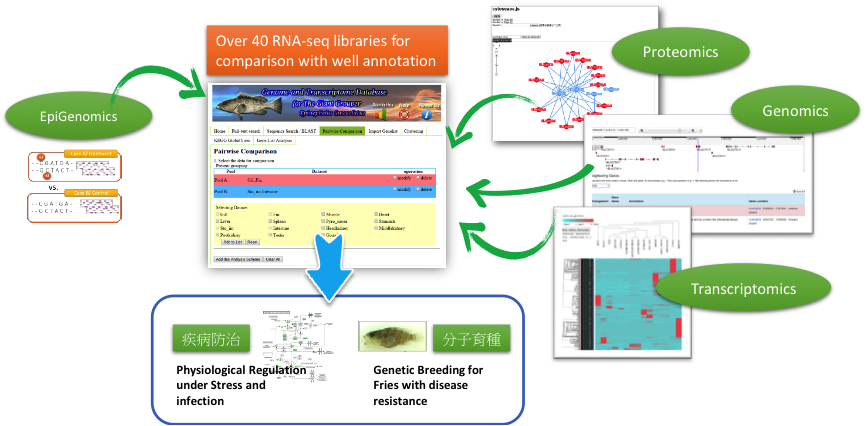
我們發展 Mapreduce 平台的前組合和後組合分析技術,以預測序列片段的重複性和定序錯誤,來優化新穎基因體組合技術的正確性和效率 (https://github.com/moneycat/QReadSelector)。此外,我們也正在發展嶄新的雲端系統架構與演算法,將結合次世代與第三代單分子定序技術所產出之巨量資料,進一步加速新穎基因體組合器的執行效率與運用範疇。在基因體組裝完成後,透過我們所建構基因結構解析註解流程、線上即時分析與呈現平台 (http://molas.iis.sinica.edu.tw),已能整合基因體、轉錄體、蛋白質體與甲基化 (http://tea.iis.sincia.edu.tw) 等多維體資料,協同研究團隊以網頁介面,透過不同的角度來詮釋複雜生命現象的核心課題,如癌症臨床樣本、非模式生物(如龍膽石斑 (http://molas.iis.sinica.edu.tw/grouper2016)、 日本 鰻 (http://molas.iis.sinica.edu.tw/jpeel2016) 與雞肉絲菇等)、環境微生物與人類病毒 (Enterovirus, HCV) 研究等,已協助找出罕見疾病之致病融合基因、建構高品質之非模式物種資料庫與找出限制 HCV 病毒複製的機制 (PNAS,2017) 等。在複雜網路關鍵因子分析上,過去整合十一種拓樸分析之程式 cytohubba (http://apps.cytoscape.org/apps/cytohubba),自 2011 年以來,已有超過 9,000 次的下載,近 180 次的文獻引用,而 2017 年一月上線的新版,至三月底,已有超過四百次的下載。此外,所發展之多維體相關演算法已開始轉換到 Galaxy 平台介面,將以 DOCKER 或是 VM 影像檔方式散佈,以期讓生物學家、臨床研究人員和生物資訊學家可以輕易地獲得與整合這些工具,加速整體的研究流程。
轉錄因子和 DNA 結構的調控關係:
轉錄因子結合的預測由 DNA 序列與染色質通透性所決定,而後者通常受到染色質狀態與 DNA 結構特性所影響。藉由同時考慮這些特徵,我們提出的隨機森林分類器在預測轉錄分子結合有顯著的改善。接下來我們將進一步針對對於任何已定序之物種,單就其 DNA 序列來正確預測轉錄因子結合的可能區域。在染色體的研究方面, 我們發現非 B 型 DNA 結構在人類與老鼠中與選擇性剪接高度關聯,顯見結構妨礙在轉錄 - 剪接中扮演重要角色。更進一步發現與 DNA 迴路有關的增強子,在老鼠中附近轉錄的 eRNA 極可能參與調控該增強子之目標基因。
(2) 蛋白體及代謝體研究
蛋白體質譜實驗大數據分析:
質譜儀已成研究蛋白體學重要的實驗技術,質譜實驗有由下而上和由上而下兩類,前者是較常用的方式;我們針對此種實驗方式發展了計算方法和工具進行蛋白體定性及定量分析。大規模帶醣的醣蛋白鑑定分析極具挑戰,故目前少有具此功能的工具。為此,我們提出計算方法,並發展名為 MAGIC 的系統。我們亦建構 MAGIC-web 的平台,處理大規模及標靶醣蛋白的鑑定。在蛋白體定量方面,我們正將前已發表的定量工具Multi-Q,重新開發,以處理目前盛行的 TMT 10-plex 定量實驗。由上而下的質譜實驗方式,能完整偵測蛋白質上的修飾,故逐漸受到重視;唯其資料分析複雜,主要難在決定蛋白質的訊號群及其電荷。我們提出一計算方法,名之為 DYAMOND, 並以此開發名為 iTop-Q 的分析軟體。
醣合成及代謝體之自動分析:
醣的合成技術雖然已臻成熟,然而複雜的實驗步驟,讓許多研究單位望之卻步。為了減少這種嘗試、錯誤的實驗方法,我們發展了一套全自動的程式,將生物化學的種種限制因素考慮進來,預測出最佳的合成序列。此外,我們在代謝體質譜資料分析上,發展定性與定量的計算方法,並完成定量工具,目前在開發定性軟體。台灣癌症登月計畫:台灣於 2016 年八月受美國癌症研究所之邀,加入國際癌症登月計畫;其中一重要目標是進行癌症的蛋白基因體表徵分析。我們針對早發及早期的肺癌進行研究,目前已知應和一些蛋白質的變異有關。但從質譜大數據可靠地鑑定變異胜肽相當困難;我們目前正為此設計演算法。我們也發展工具,幫助研究者選擇合適的水解酶分解蛋白質,以利產生合適長度的胜肽,質譜產生較好的訊號,而較有機會偵測變異胜肽。此計畫採用 TMT 10-plex 標記進行定量實驗,我們發展中的新版 Multi-Q 2 軟體,將用來定量分析。我們已展開肺癌病人檢體的質譜實驗數據分析,依美國癌症研究所的品管要求,每組病人資料必須鑑定出 1 萬個蛋白質,我們已能達到此要求。同時,我們亦將針對這些質譜大數據進行變異胜肽的鑑定。
與國內外的合作單位:
生物資訊研究是跨領域研究,我們研究合作夥伴包含本院的農生中心、生物多樣性中心、化學所、統計所、分子生物研究所、細胞生物與個體生物研究所、植微所、和生醫所等,及院外的國衛院、台灣大學(醫學院、藥理所、病理研究所、生科院、漁科所和農業化學系)、台大醫院、成功大學生命科學院、海洋大學水產養殖系和水產試驗所等。另外與國外的學術單位也有密切的合作計畫進行,包含美國密西根州立大學植物生物學研究所、加州大學洛杉磯分校醫學院、日本大阪大學蛋白質研究所和日本產業技術總合研究所等。
